文章题目:Contrasting patterns of antibiotic resistance and metal resistance genes between free living and particle attached bacteria in a hypertrophic lake
期刊信息:Environmental Technology & Innovation (JCR一区,IF=7.1)
内容简介:
人类活动引起的富营养化和藻华显著改变了湖泊微生物群落的结构。然而,这一过程是否也会导致抗生素耐药基因(ARGs)、金属耐药基因(MRGs)和可移动遗传元件(MGEs)的传播发生变化,亟需研究。本研究采用宏基因组方法,研究蓝藻藻华期间自由生活(FL)和颗粒相关(PA)组分中ARGs、MRGs、MGEs和毒力因子(VFs)的丰度、多样性、关系及宿主。FL组的抗性基因多样性显著更高,而PA组的MRGs和MGEs丰度更高。大型蓝藻聚集体可能改变细菌群落结构,调节不同组分间抗药基因的多样性和丰度。这些聚集体增强了微生物网络的拓扑复杂性和多维连接性,同时提升了转座子丰度。宏基因组组装结果显示,转座子与MRGs在蓝藻衍生的组装序列中高频共现。这表明富含转座酶的蓝藻聚集体可能促进MRGs在环境中的传播。此外,我们还发现了携带多种耐药基因和VF的潜在致病细菌。本研究系统地分析了蓝藻藻华期间不同组分的抗性基因,揭示了ARGs与MRGs之间的明显趋势。这些发现为制定有效的湖泊管理策略以遏制抗性基因的传播提供了关键见解。
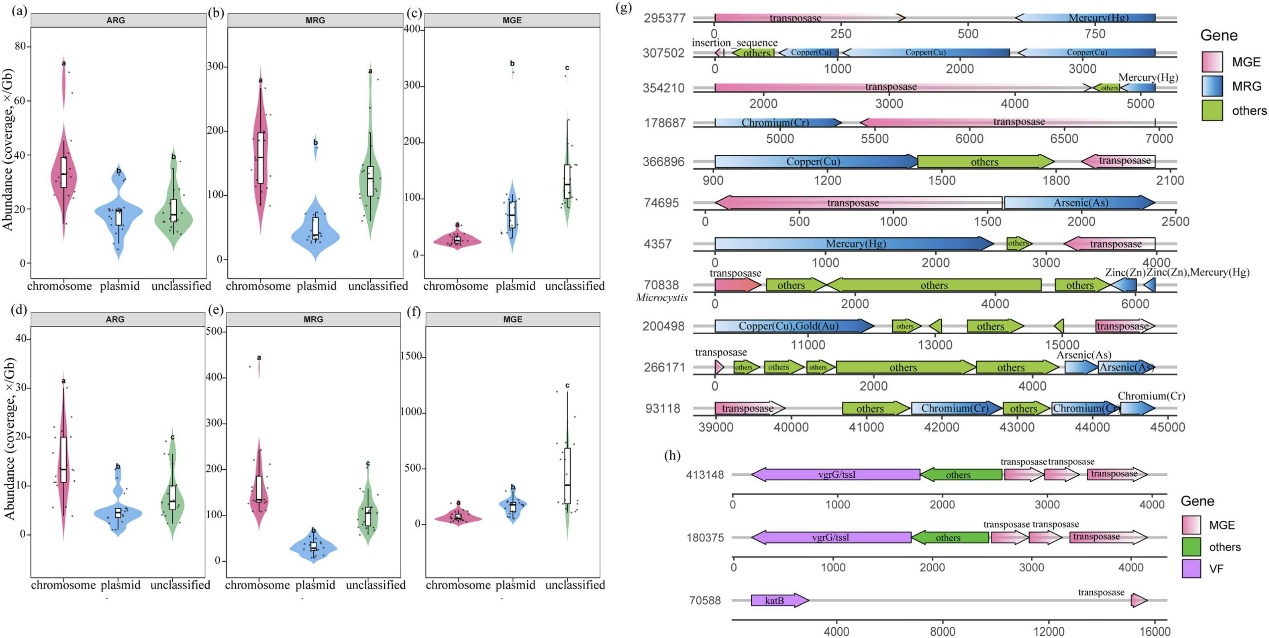
图1. FL和PA组分中ARG、MRGs和MGE的遗传位置及其共现情况。小提琴图展示了FL(a)和PA(d)染色体和质粒上的ARGs丰度。FL(b)和PA(e)染色体和质粒上的MRGs丰度。FL(c)和PA(f)染色体和质粒上的MGE丰度。(g)MRGs和转座酶在共享基因组系的遗传共定位。(h)基因组系中VFs和MGEs的共定位。
结论:
在蓝藻高峰期,FL细菌群落携带更丰富的ARGs、MRGs和MGEs,而PA细菌群落则表现出更高的MRGs和MGE丰度。大型蓝藻聚集体显著改变了细菌群落结构,调节了不同组分中耐药基因的多样性和丰度。这些聚集体增强了微生物网络的拓扑复杂性和多维连接性,同时提升了转座子丰度。这种升高的转座子丰度可能促进MRGs的水平传递,以及携带多药耐药基因和VFs的临床相关菌株的传播。鉴于湖泊微生物生态系统中选择压力日益复杂,研究耐药基因在强烈微生物波动期间不同组分间的潜在传递途径和分布模式至关重要。
该成果以“Contrasting patterns of antibiotic resistance and metal resistance genes between free living and particle attached bacteria in a hypertrophic lake”发表在国际环境领域著名期刊《Environmental Technology & Innovation》,全球变化生态学实验室硕士生王盛兴为第一作者,万师强教授为共同作者,中国科学院南京地理与湖泊研究所吴庆龙研究员与周丽君副研究员为共同通讯作者。

